genetica e
trasmissione daltonismo associato a cromosoma X
o con ipotesi se fosse associato a cromosoma somatico
trasmissione
daltonismo associato a cromosoma X
(o se fosse associato a cromosoma somatico)
http://www.youtube.com/watch?v=1u14NJB6nM0

program va1; (* variante di varia1 *)
uses crt,graph;
procedure grafica;
var t,s:integer;
stringa:string;
begin
t:=0;
s:=0;
stringa:=('c:\SCHEDA\');
initgraph(s,t,stringa);
end;
procedure attende;
begin
delay(2000);
end;
procedure pausa1;
begin
setcolor(4);
outtextxy(430,10,'premi INVIO');readln;
setcolor(0);
outtextxy(430,10,'premi INVIO');
setcolor(15);
end;
procedure pausa;
begin
readln;cleardevice;
end;
procedure testo(x,y:integer;st:string);
begin
outtextxy(x,y,st);
end;
procedure ma(x1,y1,x2,y2,st,cm:integer);
begin
setfillstyle(st,cm);
bar(x1,y1,x2,y2);
end;
procedure fe(x1,y1,rv,ro,st,cf:integer);
begin
setfillstyle(st,cf);
fillellipse(x1,y1,rv,ro);
end;
procedure programma;
begin
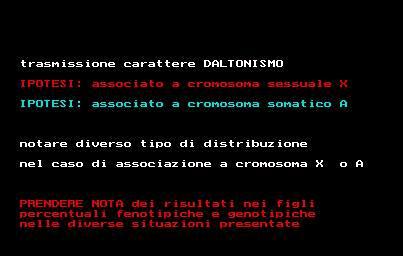
outtextxy(20,60,'trasmissione carattere DALTONISMO ');
setcolor(4);
outtextxy(20,80,'IPOTESI: associato a cromosoma sessuale X ');
setcolor(3);
outtextxy(20,100,'IPOTESI: associato a cromosoma somatico A ');
setcolor(15);
outtextxy(20,140,'notare diverso tipo di distribuzione');
outtextxy(20,160,'nel caso di associazione a cromosoma X o A ');
setcolor(4);
outtextxy(20,200,'PRENDERE NOTA dei risultati nei figli');
outtextxy(20,210,'percentuali fenotipiche e genotipiche');
outtextxy(20,220,'nelle diverse situazioni presentate ');
setcolor(15);
pausa;
end;
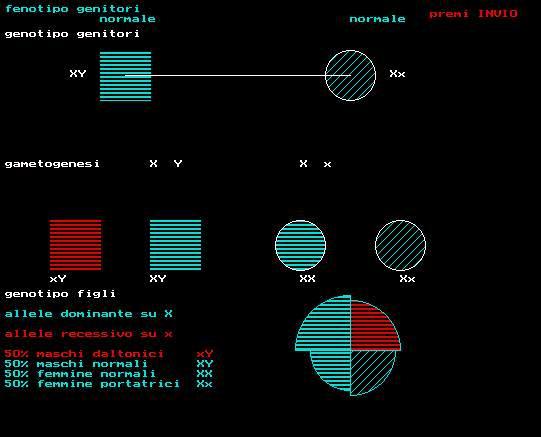
procedure pro1;
begin
(* associato a X *)
ma(100,50,150,100,2,3);
fe(350,75,25,25,2,3);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,2,3);
setcolor(3);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'normale');
outtextxy(350,15,'normale');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'XY');
outtextxy(390,70,'Xx');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'X Y');
outtextxy(300,160,'X x');
setcolor(3);outtextxy(5,310,'allele dominante su X ');
setcolor(4);outtextxy(5,330,'allele recessivo su x ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'xY');
outtextxy(150,275,'XY');
outtextxy(300,275,'XX');
outtextxy(400,275,'Xx');
attende;
fe(350,75,25,25,3,3);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,3,3);attende;
setcolor(4);
outtextxy(5,350,'50% maschi daltonici xY ');
setcolor(3);
outtextxy(5,360,'50% maschi normali XY ');
outtextxy(5,370,'50% femmine normali XX ');
outtextxy(5,380,'50% femmine portatrici Xx ');
setfillstyle(2,4);pieslice(350,350,0,90,50);
setfillstyle(2,3);pieslice(350,350,90,180,55);
setfillstyle(2,3);pieslice(350,350,180,270,40);
setfillstyle(3,3);pieslice(350,350,270,360,45);
setcolor(15);
pausa1;cleardevice;
end;
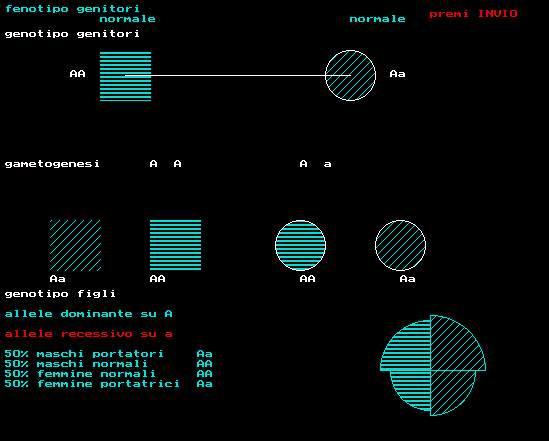
procedure pro2;
begin
(* associato a cromosoma A *)
ma(100,50,150,100,2,3);
fe(350,75,25,25,2,3);
line(125,75,350,75);
ma(50,220,100,270,2,3);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,2,3);
setcolor(3);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'normale');
outtextxy(350,15,'normale');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'AA');
outtextxy(390,70,'Aa');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'A A');
outtextxy(300,160,'A a');
setcolor(3);outtextxy(5,310,'allele dominante su A ');
setcolor(4);outtextxy(5,330,'allele recessivo su a ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'Aa');
outtextxy(150,275,'AA');
outtextxy(300,275,'AA');
outtextxy(400,275,'Aa');attende;
fe(350,75,25,25,3,3);
line(125,75,350,75);
ma(50,220,100,270,3,3);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,3,3);attende;
setcolor(3);
outtextxy(5,350,'50% maschi portatori Aa ');
outtextxy(5,360,'50% maschi normali AA ');
outtextxy(5,370,'50% femmine normali AA ');
outtextxy(5,380,'50% femmine portatrici Aa ');
attende;
setfillstyle(3,3);pieslice(430,370,0,90,55);
setfillstyle(2,3);pieslice(430,370,90,180,50);
setfillstyle(2,3);pieslice(430,370,180,270,40);
setfillstyle(3,3);pieslice(430,370,270,360,45);attende;
setcolor(15);
pausa1;cleardevice;
end;
procedure pro3;
begin
(* associato a X *)
ma(100,50,150,100,2,4);
fe(350,75,25,25,2,3);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,2,4);
setcolor(3);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'daltonico');
outtextxy(350,15,'normale');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'xY');
outtextxy(390,70,'xX');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'x Y');
outtextxy(300,160,'x X');
setcolor(3);outtextxy(5,310,'allele dominante su X ');
setcolor(4);outtextxy(5,330,'allele recessivo su x ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'xY');
outtextxy(150,275,'XY');
outtextxy(300,275,'xX');
outtextxy(400,275,'xx');
attende;
fe(350,75,25,25,3,3);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,3);
fe(300,245,25,25,3,3);
fe(400,245,25,25,2,4);
setcolor(4);
outtextxy(5,350,'50% maschi daltonici xY ');
outtextxy(5,360,'50% femmine daltoniche xx ');
setcolor(3);
outtextxy(5,370,'50% maschi normali XY ');
outtextxy(5,380,'50% femmine portatrici Xx ');
setfillstyle(2,4);pieslice(430,370,0,90,55);
setfillstyle(2,4);pieslice(430,370,90,180,50);
setfillstyle(2,3);pieslice(430,370,180,270,40);
setfillstyle(3,3);pieslice(430,370,270,360,45);attende;
setcolor(15);
pausa1;cleardevice;
end;
procedure pro4;
begin
(* associato ad A *)
ma(100,50,150,100,2,4);
fe(350,75,25,25,2,3);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,2,4);
setcolor(3);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'daltonico');
outtextxy(350,15,'normale');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'aa');
outtextxy(390,70,'Aa');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'a a');
outtextxy(300,160,'a A');
setcolor(3);outtextxy(5,310,'allele dominante su A ');
setcolor(4);outtextxy(5,330,'allele recessivo su a ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'aa');
outtextxy(150,275,'Aa');
outtextxy(300,275,'Aa');
outtextxy(400,275,'aa');
attende;
fe(350,75,25,25,3,3);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,3,3);
fe(300,245,25,25,3,3);
fe(400,245,25,25,2,4);attende;
setcolor(4);
outtextxy(5,350,'50% maschi daltonici aa ');
outtextxy(5,360,'50% femmine daltoniche aa ');
setcolor(3);
outtextxy(5,370,'50% maschi portatori Aa ');
outtextxy(5,380,'50% femmine portatrici Aa ');
setfillstyle(2,4);pieslice(430,370,0,90,55);
setfillstyle(2,4);pieslice(430,370,90,180,50);
setfillstyle(3,3);pieslice(430,370,180,270,40);
setfillstyle(3,3);pieslice(430,370,270,360,45);attende;
setcolor(15);
pausa1;cleardevice;
end;
procedure pro5;
begin
(* associato a X *)
ma(100,50,150,100,2,4);
fe(350,75,25,25,2,3);
line(125,75,350,75);
ma(50,220,100,270,2,3);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,2,3);
setcolor(3);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'daltonico');
outtextxy(350,15,'normale');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'xY');
outtextxy(390,70,'XX');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'x Y');
outtextxy(300,160,'X X');
setcolor(3);outtextxy(5,310,'allele dominante su X ');
setcolor(4);outtextxy(5,330,'allele recessivo su x ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'XY');
outtextxy(150,275,'XY');
outtextxy(300,275,'Xx');
outtextxy(400,275,'Xx');
attende;
ma(50,220,100,270,2,3);
ma(150,220,200,270,2,3);
fe(300,245,25,25,3,3);
fe(400,245,25,25,3,3);
setcolor(3);
outtextxy(5,350,'100% maschi normali XY ');
outtextxy(5,360,'100% femmine portatrici xX ');
setfillstyle(2,3);pieslice(430,370,0,180,50);
setfillstyle(3,3);pieslice(430,370,180,360,45);
setcolor(15);
pausa1;cleardevice;
end;
procedure pro6;
begin
(* associato a A *)
ma(100,50,150,100,2,4);
fe(350,75,25,25,2,3);
line(125,75,350,75);
ma(50,220,100,270,2,3);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,2,3);
setcolor(3);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'daltonico');
outtextxy(350,15,'normale');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'aa');
outtextxy(390,70,'AA');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'a a');
outtextxy(300,160,'A A');
setcolor(3);outtextxy(5,310,'allele dominante su A ');
setcolor(4);outtextxy(5,330,'allele recessivo su a ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'Aa');
outtextxy(150,275,'Aa');
outtextxy(300,275,'Aa');
outtextxy(400,275,'Aa');attende;
ma(50,220,100,270,3,3);
ma(150,220,200,270,3,3);
fe(300,245,25,25,3,3);
fe(400,245,25,25,3,3);
setcolor(3);
outtextxy(5,350,'100% maschi portatori Aa ');
outtextxy(5,360,'100% femmine portatrici Aa');
setfillstyle(3,3);pieslice(430,370,0,180,50);
setfillstyle(3,3);pieslice(430,370,180,360,45);
setcolor(15);
pausa1;cleardevice;
end;
procedure pro7;
begin
(* associato a X *)
ma(100,50,150,100,2,4);
fe(350,75,25,25,2,4);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,4);
fe(300,245,25,25,2,4);
fe(400,245,25,25,2,4);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'daltonico');
outtextxy(350,15,'daltonico');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'xY');
outtextxy(390,70,'xx');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'x Y');
outtextxy(300,160,'x x');
setcolor(3);outtextxy(5,310,'allele dominante assente');
setcolor(4);outtextxy(5,330,'allele recessivo su x ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'xY');
outtextxy(150,275,'xY');
outtextxy(300,275,'xx');
outtextxy(400,275,'xx');
setcolor(4);
outtextxy(5,350,'100% maschi daltonici xY ');
outtextxy(5,360,'100% femmine daltoniche xx ');
setfillstyle(2,4);pieslice(430,370,0,180,50);
setfillstyle(2,4);pieslice(430,370,180,360,45);
attende;setcolor(15);
pausa1;cleardevice;
end;
procedure pro8;
begin
(* associato ad A *)
ma(100,50,150,100,2,4);
fe(350,75,25,25,2,4);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,4);
fe(300,245,25,25,2,4);
fe(400,245,25,25,2,4);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'daltonico');
outtextxy(350,15,'daltonico');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'aa');
outtextxy(390,70,'aa');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'a a');
outtextxy(300,160,'a a');
setcolor(3);outtextxy(5,310,'allele dominante assente');
setcolor(4);outtextxy(5,330,'allele recessivo su x ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'aa');
outtextxy(150,275,'aa');
outtextxy(300,275,'aa');
outtextxy(400,275,'aa');
setcolor(4);
outtextxy(5,350,'100% maschi daltonici aa ');
outtextxy(5,360,'100% femmine daltoniche aa ');
setfillstyle(2,4);pieslice(430,370,0,180,50);
setfillstyle(2,4);pieslice(430,370,180,360,45);
setcolor(15);
pausa1;cleardevice;
end;
procedure pro9;
begin
(* associato a X *)
ma(100,50,150,100,2,3);
fe(350,75,25,25,2,4);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,4);
fe(300,245,25,25,2,3);
fe(400,245,25,25,2,3);
setcolor(15);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'normale');
outtextxy(350,15,'daltonico');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'XY');
outtextxy(390,70,'xx');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'X Y');
outtextxy(300,160,'x x');
setcolor(3);outtextxy(5,310,'allele dominante su X ');
setcolor(4);outtextxy(5,330,'allele recessivo su x ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'xY');
outtextxy(150,275,'xY');
outtextxy(300,275,'Xx');
outtextxy(400,275,'Xx');attende;
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,4);
fe(300,245,25,25,3,3);
fe(400,245,25,25,3,3);attende;
setcolor(4);
outtextxy(5,350,'100% maschi daltonici xY ');
setcolor(3);
outtextxy(5,370,'100% femmine portatrici Xx ');
setfillstyle(2,4);pieslice(430,370,0,180,50);
setfillstyle(3,3);pieslice(430,370,180,360,45);
setcolor(15);
pausa1;cleardevice;
end;
procedure pro10;
begin
ma(100,50,150,100,2,3);
fe(350,75,25,25,2,4);
line(125,75,350,75);
ma(50,220,100,270,2,4);
ma(150,220,200,270,2,3);
fe(300,245,25,25,2,3);
fe(400,245,25,25,2,4);
setcolor(15);
outtextxy(5,5,'fenotipo genitori');
outtextxy(100,15,'normale');
outtextxy(350,15,'daltonico');
attende;
setcolor(15);
outtextxy(5,30,'genotipo genitori');
outtextxy(70,70,'Aa');
outtextxy(390,70,'aa');
outtextxy(5,160,'gametogenesi');
outtextxy(150,160,'A a');
outtextxy(300,160,'a a');
setcolor(3);outtextxy(5,310,'allele dominante su A ');
setcolor(4);outtextxy(5,330,'allele recessivo su a ');
setcolor(15);outtextxy(5,290,'genotipo figli');
outtextxy(50,275,'aa');
outtextxy(150,275,'Aa');
outtextxy(300,275,'Aa');
outtextxy(400,275,'aa');attende;
ma(50,220,100,270,2,4);
ma(150,220,200,270,3,3);
fe(300,245,25,25,3,3);
fe(400,245,25,25,2,4);attende;
setcolor(4);
outtextxy(5,350,'50% maschi daltonici aa ');
outtextxy(5,360,'50% femmine daltoniche aa ');
setcolor(3);
outtextxy(5,370,'50% maschi portatori Aa');
outtextxy(5,380,'50% femmine portatrici Aa');
setfillstyle(2,4);pieslice(430,370,0,90,55);
setfillstyle(2,4);pieslice(430,370,90,180,50);
setfillstyle(4,3);pieslice(430,370,180,270,40);
setfillstyle(4,3);pieslice(430,370,270,360,45);attende;
setcolor(15);
pausa1;cleardevice;
end;
procedure scelta;
var sce:integer;
begin
setcolor(4);
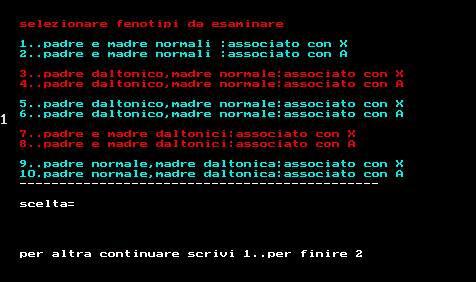
testo(20,20,'selezionare fenotipi da esaminare ');
setcolor(3);
testo(20,40,'1..padre e madre normali :associato con X ');
testo(20,50,'2..padre e madre normali :associato con A ');
setcolor(4);
testo(20,70,'3..padre daltonico,madre normale:associato con X');
testo(20,80,'4..padre daltonico,madre normale:associato con A');
setcolor(3);
testo(20,100,'5..padre daltonico,madre normale:associato con X ');
testo(20,110,'6..padre daltonico,madre normale:associato con A');
setcolor(4);
testo(20,130,'7..padre e madre daltonici:associato con X');
testo(20,140,'8..padre e madre daltonici:associato con A');
setcolor(3);
testo(20,160,'9..padre normale,madre daltonica:associato con X');
testo(20,170,'10.padre normale,madre daltonica:associato con A');
setcolor(15);
testo(20,180,'---------------------------------------------');
testo(20,200,'scelta=');readln(sce);cleardevice;
case sce of
1:pro1;2:pro2;3:pro3;
4:pro4;5:pro5;6:pro6;7:pro7;8:pro8;9:pro9;10:pro10;
end;
testo(20,250,'per altra continuare scrivi 1..per finire 2 ');readln(sce);
if sce=1 then scelta;
end;
begin
grafica;
programma; (* scrivere nome procedura propria *)
scelta;
closegraph;
end.